Továbbra is a baktérium ostorok evolúciójáról szóló cikkekből szemezgetünk. Az mai mű címe magáért beszél: A baktérium ostor lépésenkénti kialakulása.
A szerzők nem kevesebbre vállakoztak, mint hogy minden hozzáférhető baktérium genomból (41 genomról van szó, azért jegyezzük meg, hogy a 2005 -ben rendelkezésre álló 249 genomból íródott a közlemény, minden egyes nagyobb baktériumcsoportból egy gemonszekvenciát választottak ki) kikeresték az ostort felépítő fehérjéket kódoló géneket és ezek összehasonlításával próbálták meghatározni az ostor eredetét. Az első megállapításuk, hogy az ostor szerkezeti elemei minden vizsgált baktériumcsoportban jelen vannak. Ez azt jelenti, hogy egy ős-ostor már a főbb baktériumcsoportok szétválása előtt is jelen volt, ez a huszonnégy gén terméke által felépített „mag” minden baktérium ostor közös őséből származik (ezek az ábrán kékkel jelöltek). Azért ezek sem teljesen univerzálisak, a Thermotoga maritima nevű jószágban nem működő pszeudogén az flgE, a Listeria innocua nevű jószágból hiányzik az flgF gén, az fliQ pedig Clostridium tetaniban nem található meg. Ezen kívül például a Spirochétákból hiányzik az FlgH és FlgI gén, mivel az ős ő ostoruk nem nyúlik át a külső membránon.
Továbbra is a baktérium ostorok evolúciójáról szóló cikkekből szemezgetünk. Az mai mű címe magáért beszél: A baktérium ostor lépésenkénti kialakulása.
A szerzők nem kevesebbre vállakoztak, mint hogy minden hozzáférhető baktérium genomból (41 genomról van szó, azért jegyezzük meg, hogy a 2005 -ben rendelkezésre álló 249 genomból íródott a közlemény, minden egyes nagyobb baktériumcsoportból egy gemonszekvenciát választottak ki) kikeresték az ostort felépítő fehérjéket kódoló géneket és ezek összehasonlításával próbálták meghatározni az ostor eredetét. Az első megállapításuk, hogy az ostor szerkezeti elemei minden vizsgált baktériumcsoportban jelen vannak. Ez azt jelenti, hogy egy ős-ostor már a főbb baktériumcsoportok szétválása előtt is jelen volt, ez a huszonnégy gén terméke által felépített „mag” minden baktérium ostor közös őséből származik (ezek az ábrán kékkel jelöltek). Azért ezek sem teljesen univerzálisak, a Thermotoga maritima nevű jószágban nem működő pszeudogén az flgE, a Listeria innocua nevű jószágból hiányzik az flgF gén, az fliQ pedig Clostridium tetaniban nem található meg. Ezen kívül például a Spirochétákból hiányzik az FlgH és FlgI gén, mivel az ős ő ostoruk nem nyúlik át a külső membránon.
 A szerzők csak a huszonnégy magfehérje történetének a felvázolására vállalkoztak, így csak ezekről lesz szó. Először egy leszármazási fát szerkesztettek a gének különböző baktériumokból származó ortológjaiból, majd ezt összehasonlították tizennégy ostorfehérje génjéből képzett leszármazási fával. Minden egyes gén illesztése pontosan ugynazt a leszármazási fát eredményezte, mint amit a tizennégy fehérje vizsgálatával kaptak, így megállapíthatták, hogy a vizsgált magi gének evolúciós története azonos, együtt változtak a keletkezésük óta. Ezek után az egyes gének leszármazási fáját (B fa) összehasonlították másik huszonöt, az ostorhoz semmiben sem kapcsolódó gén leszármazási fájával (A fa), hogy kimutathatóak legyenek az esetleges horizontális géntranszfer események. Két ilyen esemény nyomára bukkantak (bekeretezve), az egyik a Zymomonas nobilis nevű jószág, a másik pedig három közeli rokon Betaproteobacteria faj, ezek nyilván horizontális géntranszferrel jutottak az ostorfehérjéket kódoló génjeikhez.
A szerzők csak a huszonnégy magfehérje történetének a felvázolására vállalkoztak, így csak ezekről lesz szó. Először egy leszármazási fát szerkesztettek a gének különböző baktériumokból származó ortológjaiból, majd ezt összehasonlították tizennégy ostorfehérje génjéből képzett leszármazási fával. Minden egyes gén illesztése pontosan ugynazt a leszármazási fát eredményezte, mint amit a tizennégy fehérje vizsgálatával kaptak, így megállapíthatták, hogy a vizsgált magi gének evolúciós története azonos, együtt változtak a keletkezésük óta. Ezek után az egyes gének leszármazási fáját (B fa) összehasonlították másik huszonöt, az ostorhoz semmiben sem kapcsolódó gén leszármazási fájával (A fa), hogy kimutathatóak legyenek az esetleges horizontális géntranszfer események. Két ilyen esemény nyomára bukkantak (bekeretezve), az egyik a Zymomonas nobilis nevű jószág, a másik pedig három közeli rokon Betaproteobacteria faj, ezek nyilván horizontális géntranszferrel jutottak az ostorfehérjéket kódoló génjeikhez.
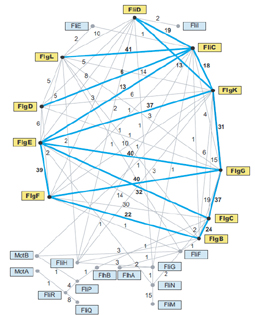 Ezek után megvizsgálták, hogy mely fehérjékehez hasonlítanak legjobban az ostorfehérjék, minden egyes vizsgált baktérium genom minden egyes fehérjéjéhez illesztve őket, azt találták, hogy leginkább egymáshoz, így vélhetőleg egymásból származnak. A hozzájuk leginkább hasonló fehérjék a pílusfehérjék és az V típusú szekréciós rendszer fehérjéi, valamint néhány membránfehérje. Az elemzés megerősítette a fehérjék rokonságát, az fliN és fliM fehérjék számos baktériumcsoportban egymás közeli homológjai, ugyanígy, ha nem csak az E. coli genomot vizsgálták, számos csoportban az fliP, fliR, fliQ fehérjék erős homológiát mutattak. Ezekkel az összehasonlításokkal sikerült tisztázni a vizsgált gének leszármazását. Erről készítettek egy bevallottan bonyolult ábrát is, ahol azt ábrázolták, hogy hány genomban mutatott erős homológiát egymáshoz két fehérje. A vastag kék vonalak azt jelzik, az E. coli fehérjekomplex tagjai között milyen homológiaviszonyok mutathatóak ki, a vékony szürke vonalak pedig azokat a fehérjéket jelölik, amelyek valamelyik másik baktériumban egymás homológjai, a számok pedig azt mutatják, hány genomban mutatható ki a két fehérje között homológia. Ha megnézzük az ábrát, éppen az tűnhet föl elsőre, hogy azok a fehérjék, amik E. coliban erős homológiát mutatnak, korántsem ilyen erős homológok az összes többi vizsgált baktériumban. Nyilván ez úgy magyarázható, hogy egy közös ősi ostorból kiindulva az egyes baktériumcsoportokban más-más irányba változtak az egyes fehérjék.
Ezek után megvizsgálták, hogy mely fehérjékehez hasonlítanak legjobban az ostorfehérjék, minden egyes vizsgált baktérium genom minden egyes fehérjéjéhez illesztve őket, azt találták, hogy leginkább egymáshoz, így vélhetőleg egymásból származnak. A hozzájuk leginkább hasonló fehérjék a pílusfehérjék és az V típusú szekréciós rendszer fehérjéi, valamint néhány membránfehérje. Az elemzés megerősítette a fehérjék rokonságát, az fliN és fliM fehérjék számos baktériumcsoportban egymás közeli homológjai, ugyanígy, ha nem csak az E. coli genomot vizsgálták, számos csoportban az fliP, fliR, fliQ fehérjék erős homológiát mutattak. Ezekkel az összehasonlításokkal sikerült tisztázni a vizsgált gének leszármazását. Erről készítettek egy bevallottan bonyolult ábrát is, ahol azt ábrázolták, hogy hány genomban mutatott erős homológiát egymáshoz két fehérje. A vastag kék vonalak azt jelzik, az E. coli fehérjekomplex tagjai között milyen homológiaviszonyok mutathatóak ki, a vékony szürke vonalak pedig azokat a fehérjéket jelölik, amelyek valamelyik másik baktériumban egymás homológjai, a számok pedig azt mutatják, hány genomban mutatható ki a két fehérje között homológia. Ha megnézzük az ábrát, éppen az tűnhet föl elsőre, hogy azok a fehérjék, amik E. coliban erős homológiát mutatnak, korántsem ilyen erős homológok az összes többi vizsgált baktériumban. Nyilván ez úgy magyarázható, hogy egy közös ősi ostorból kiindulva az egyes baktériumcsoportokban más-más irányba változtak az egyes fehérjék.
Az ebből a vizsgálatból levonható következtetések egyszerűek: Az ősibb fehérjék a sejtmembránba ágyazott egységet felépítők, a fiatalabbak az ostor külső egységeit alkotók. Mivel a legősibb fehérjéi még felismerhető homológjai az ATPáz béta alegységének (FliI) és a TolQ-TolR szekréciós fehérjéknek (MotA, MotB), feltételezhető, hogy az ostor őse egy membránba ágyazott szekréciós fehérjekomplex volt, valamint a forgómozgást végző ATPáz komplex. Később ehhez az alaphoz csatlakozott a tulajdonképpeni ostort alkotó többi fehérje, amik mind az ősi néhány szekvenciából jöttek létre dupikációkkal, majd mutációkkal.
Bár azért mindig szem előtt kell tartani, hogy az ilyen in silico szekvenciaelemzések eredményét érdemes egészséges óvatossággal kezelni, ez egy nagyon szép példája annak, hogy a nagyon sok genomot felhasználó összehasonlító elemzések olyan összefüggéseket is megvilágíthatnak, amik egyetlen szervezet, például az E. coli vizsgálatakor rejtve maradnak, valamint egy független módszer is visszaigazolja, hogy az ostor kialakulásra vonatkozó feltételezéseink helyesek, egy egyszerű szekréciós rendszer és egy forgó mozgást végző ATPáz kapcsolódásával jött létre az a kezdetleges fehérjekomplex, ami a mai baktérium ostorok ősének tekinthető.
Nekem a cikk olvasása után még két kérdésem maradt, az első, hogy miért nem találták nyomát a legutóbb tárgyalthoz hasonló hibrid flagellumoknak, bár a válasz valószínűleg annyi, hogy negyven olyan baktériumot választottak ki, ahol pont nem történt hasonló eset. A másik, hogy mi van azokkal a szervezetekkel, amelyeknek két különböző flagelluma is van, de ilyet nem tárgyalnak, gondolom 2005 –ben még nem állt rendelkezésükre ilyen teljes genom, vagy ilyet eleve nem vettek be a vizsgálatba.
Renyi Liu and Howard Ochman (2007): Stepwise formation of the bacterial flagellar system PNAS vol. 104 no. 17 7116–7121
Sexcomb