A humán és egyéb genom projekteknek köszönhetően most már egyre pontosabb becsléseink vannak arra vonatkozóan, hogy egy élőlény génállományának mekkora része tartalmaz fehérje-kódoló géneket. Ez amúgy meghökkentően kevés, az ember esetében mindössze 1,4%. Hogy a többi DNS mit is csinál? Jó kérdés. Egy részük például szabályozza a gének kifejeződését.
A nem kódoló DNS egy része cisz-elem, definíció szerint az ugyanazon DNS-szálon lévő gén működését szabályozó szekvencia. Tipikusan ide sorolandók a promóterek, enhanszerek, inzulátorok és silencerek (nem és nem, ezt nem vagyok hajlandó fonetikusan kiírni). A másik típusba a transz-szabályozó elemek tartoznak (például a transzkripciós faktor gének), az ezekről átíródó fehérjék pozitív illetve negatív irányban befolyásolják a génexpressziót. Ne higgyük azonban, hogy csak fehérjék működhetnek transz-szabályozó elemként. A mikroRNSek (miRNSek) 20-22 bázispár hosszú, egyszálú RNS molekulák, amelyek képesek a velük komplementer szakaszt tartalmazó mRNSekhez kapcsolódva gátolni az azokról történő fehérjeszintézist és aktiválni az mRNS lebontását végző fehérje komplexet (Argonaute - AGO). Bár a szabályozás jelen esetben más szinten folyik, mint a transzkripciós faktoroknál (a TF-ok az mRNS átírása előtt, a génen fejtik ki hatásukat, míg a miRNS-ek az átírt mRNS mennyiségét, életidejét és „felhasználását” irányítják), a két csoport sok tulajdonságban hasonlít egymásra [1].
Emberben eddig közel 1000 miRNS-t írtak le, melyek a sejtdifferenciációtól az immunrendszeren keresztül a rákos transzformációig számos élettani folyamat szabályzásában vesznek részt [2]. Arról azonban, hogy az mRNS-ek hogyan menekülhetnek meg a miRNS-ek elől, kevés az információnk.
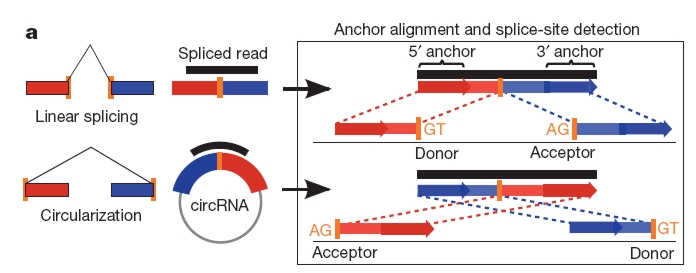
Na itt kapcsolódnak be a történetbe a címben szereplő, ezidáig ritkának hitt kör alakú RNSek (circRNSek). Nigro és társai 1991-ben egy abnormális transzkriptről számoltak be, amelyben a splicing során az eredetileg upstream exon első nukleotidja az eredetileg downstream exon utolsó nukleotidjához kapcsolódott, tehát az exonok sorrendje a génben található sorrendhez képest felcserélődött. Mindezt az alternatív splicing számlájára írták, és elkönyvelték nagy ritkaságnak [3]. Aztán 1993-ban kiderült, hogy a szex-determinációban fontos szerepet játszó SRY génről az egér herében is egy ilyen furcsa, a splicing során összekevert RNS íródik át, ami ráadásul kör alakú [4].
Múlt héten azonban több együttműködő berlini kutatócsoport közölte le több ezer (majdnem kétezer emberi, körülbelül ugyanennyi egér, és több, mint hétszáz C. elegans) cirkuláris RNS számítógépes izolálásáról és azok közül néhány kísérletes karakterizálásáról szóló eredményét a Nature online kiadásában. A több ezer jósolt circRNS-t RNS szekvencia adatbázisok vizsgálatával kapták, splice donor és akceptor szekvenciákra kerestek rá egy új algoritmus segítségével. Már maga az a tény, hogy a legtöbb általuk talált kör alakú RNS sejttípus- illetve fejlődési állapot-függő expressziót mutatott, valamint hogy a humán circRNS-ek 85%-a valamely ismert génre illeszkedett rá, valamilyen szabályozói szerepre utalt. Mivel a vizsgált circRNS-ek szignifikánsan több miRNS kötőhelyet tartalmaztak, mint bármely kontroll szekvencia, felmerült a gyanú, hogy ezek a molekulák esetleg szivacsként összegyűjtik a sejtben található miRNS-eket, felszabadítva ezzel a hírvivőket a miRNS-kontroll alól.
A humán CDR1 (paraneopláziás cerebellum-diszpláziával összefüggésbe hozható) génről átíródó kör alakú RNS például 74 miR-7 kötőhelyet tartalmaz. UV fénnyel keresztkötött sejtlizátumból történő immunprecipitációval kimutatták, hogy a CDR1as circRNS a mikroRNS-effektor AGO komplexhez kapcsolódik, vagyis a konszenzus szekvencia alapján jósolt miRNS-kötőhelyek közül van, amelyik aktív. Sőt, mi több, az emberi és egér agy több területén mind a miR-7, mind a CDR1as egyidejűleg kimutatható, vagyis minden tényező adott ahhoz, hogy in vivo is létrejöjjön közöttük a kapcsolat. A knockout kísérlet, amelyben a CDR1as circRNS-t „kiütik”, és úgy vizsgálják, mi történik a miR-7 miRNS-sel, sajnos csak a CDR1 géntermék kiütésével együtt lett volna lehetséges, ami valószínűleg az agyfejlődést is befolyásolta volna az egerekben. Hogy mindezt kikerüljék, a CDR1 gént egyáltalán nem tartalmazó zebrahalat használták erre a célra. A kísérlet során a CDR1as circRNS-t kódoló plazmid beinjektálása a miR-7 funkcióját semlegesítő morfolinó fenotípus-mintázatát kopírozta le: az embriók nagy részében a középagy mérete jelentősen kisebb lett a normálisnál, míg a telencephalon fejlődését a kezelés nem befolyásolta.
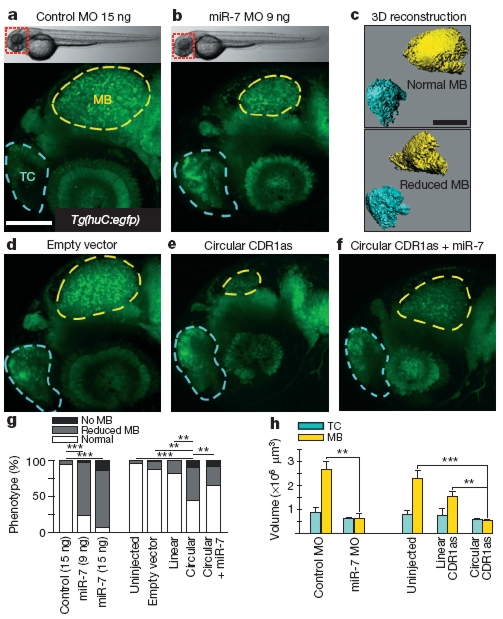
A CDR1as cirkuláris RNS tehát a miR-7 mikroRNS megkötésével annak szabályozó funkcióját gátolta a zebrahal embriókban, a középagy-fejlődésbe beleavatkozva, így igazolva a prediktált funkciót [5].
Mindez persze nem azt jelenti, hogy a cikkben prediktált összes kör alakú RNS hasonló szabályozó funkciót lát el, de mindenesetre az ismert RNS-ek világa sok izgalmas új taggal bővült, és a mikroRNS-ek szabályozásáról is nyertünk némi információt.
1. Hobert, O., Common logic of transcription factor and microRNA action. Trends Biochem Sci, 2004. 29(9): p. 462-8.
2. Bartel, D.P., MicroRNAs: target recognition and regulatory functions. Cell, 2009. 136(2): p. 215-33.
3. Nigro, J.M., et al., Scrambled exons. Cell, 1991. 64(3): p. 607-13.
4. Capel, B., et al., Circular transcripts of the testis-determining gene Sry in adult mouse testis. Cell, 1993. 73(5): p. 1019-30.
5. Memczak, S., et al., Circular RNAs are a large class of animal RNAs with regulatory potency. Nature, 2013 Feb 27. doi: 10.1038/nature11928. [Epub ahead of print]