 Az eukarióta élőlények genetikai anyaga kromoszómákba szerveződik, ez talán senkinek nem újdonság. A különböző kromoszómák száma természetesen igen változó lehet: négy darabtól néhány tucatig teljesen megszokott nagyságrendekkel állunk szemben. Az azonban, ha egy élőlénynek több ezer, noch dazu tízezernél is több kromoszómája legyen, mégiscsak figyelemreméltó.
Az eukarióta élőlények genetikai anyaga kromoszómákba szerveződik, ez talán senkinek nem újdonság. A különböző kromoszómák száma természetesen igen változó lehet: négy darabtól néhány tucatig teljesen megszokott nagyságrendekkel állunk szemben. Az azonban, ha egy élőlénynek több ezer, noch dazu tízezernél is több kromoszómája legyen, mégiscsak figyelemreméltó.
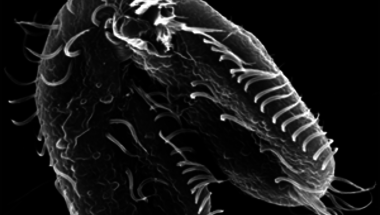
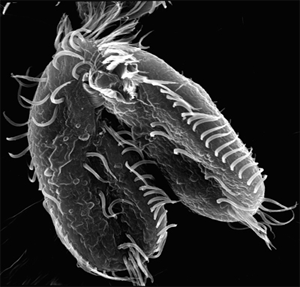
Márpedig az Oxytrichia trifallax nevű csillós egysejtű utóbbi kategóriába esik. Hogy megértsük ennek a miértjét, egy picit visszább kell lépnünk, hogy átlássuk mi mindenben fura még Oxytrichia.
Eleve a sejtekbén két sejtmag is található, egy ún. mikronukleusz (MIC) és egy makronukleusz (MAC) (ez egyébként általános a csillósok közt). A kettő között a legfeltűnöbb eltérés a méretben van: a MAC kb 2000x nagyobb és lényeges funkcionális különbség, hogy míg a MIC csak az ivaros szaporodás során játszik fontos szerepet, addig a MAC az élet egyéb területein, az ún. vegetatív életciklushoz (növekedés, táplálkozás, aszexuális szaporodás, stb.) nélkülözhetetlen.
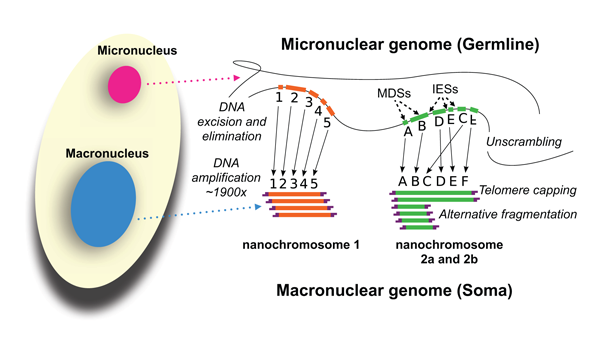
A funkcionális különbségek mögött azonban nagyon durva genetikai különbségek is vannak: míg a MIC a szokványos eukarióta exon-intron-intergénikus anyag felépítést követi, a MAC-ban gyakorlatilag mindenféle sallang nélkül fehérje kódoló régiókat találunk, nem is akárhogy: többnyire egyetlen gén egyetlen pici kromoszómát (nanokromoszómát) alkot, ahol a kódoló régiót telomérák fogják közre. (A MAC-ban bekövetkező genom-editálást annyira hatékony, az eredeti genetikai anyag nem kódoló közel 96%-a kivágódik.) S mivel a jószágnak úgy 18,500 génje lehet, ez a becslések szerint durván 16,000 kromoszómát jelent (amelyek 465 bp és 66 kb közt váltakoznak a hosszukban).
Tovább árnyalja a képet, hogy mindez a haploid becsült kormoszóma szám (mint embernél a 23) és ez egyes kromoszómák sokszoros amplifikáción átmenve akár több mint ezer (!) kópiában lehetnek jelen. Nem véletlen, hogy akkora a MAC, mint amekkora.
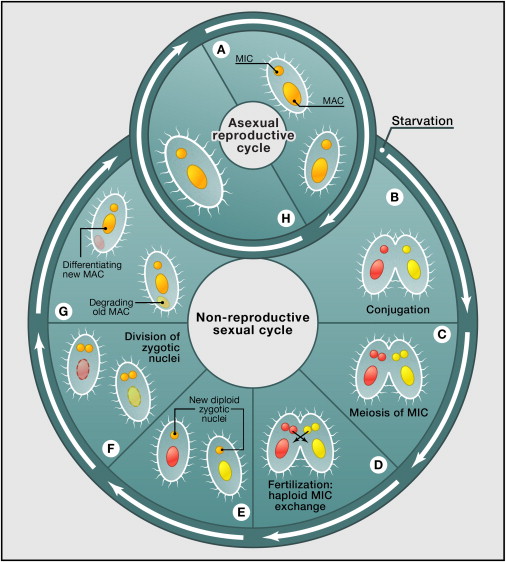 Persze a két sejtmaggal levezényelt szexuális szaporodás sem annyira egyszerű. A folyamatot (az aszexuális osztódástól eltérően) a MIC vezényli.
Persze a két sejtmaggal levezényelt szexuális szaporodás sem annyira egyszerű. A folyamatot (az aszexuális osztódástól eltérően) a MIC vezényli.
Szexuális szaporodásra akkor kerül sor, amikor hirtelen mostohává válnak a körülmények. (Ez egyébként általános stratégia a szexuális és aszexuális szaporodást váltogató élőlényeknél: a szexuális szaporodással egy sokkal diverzebb genetikai állomány jön létre, amelyben nagyobb eséllyel lesznek olyan egyedek, akik átvészelik a nehéz periódust, vagyis növelik a szülők fitneszét.)
Mivel a MIC diploid, elsőször is, akárcsak a mi ivarsejtjeink, egy számfelező sejtosztódáson, ún. meiózison kell átesnie. Ez a két Oxytrichia fúziója után következik be mindkét szülői sejtben. Majd az egyik haploid MIC mindkét szülőből a másikba vándorol, ahol fúzionál az ott maradó másik MIC-el ismét diploid sejtmagot hozva létre. Végül ez az újfent diploid MIC osztódik, és az egyik utódsejtmag az eközben lebomló MAC helyét fogja elfoglalni. És miközben ezt teszi, lezajlik a fent részletetezett drámai átrendeződés.
Hogy még cifrább legyen a helyzet, nem pusztán kivágódik egy csomó fölösleges DNS a MIC kromoszómáiból, de gyakran sorrend változások, inverziók következnek be az összevágott részek rakosgatása közben, amelyek egyrészt nagyon kiszámíthatóak, másrészt nélkülözhetetlenek, mert hiányukban nem jön létre értelmes fehérjekódoló régió.
Úgy néz ki, hogy a tervrajzot mindehez a bonyolult kirakóshoz az eredeti szülői MAC mRNS-ei biztosítják, az epigenetikai öröklődés eklatáns példájaként. Egyrészt ezek alapján fogja az őj, fejlődő MAC eldönteni, hogy melyik régiók fontosak és melyikeket kell kivágni, másrészt feltehetőleg a nukleinsav komplementaritás elve alapján a kialakuló RNS/kettősszálú-DNS hibrid fogja indukálni azokat a DNS-en belüli átrendeződéseket, amelyek a működőképes nanokromoszómákat hozzák majd létre.
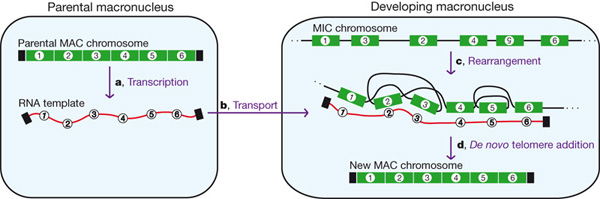
A közelmúltban leközölt MAC genomszekvenálás eredményei közt még kettő kívánkozik ide: egyrészt, a genomban számos "domesztikált" transzpozon található, amelyeknek azért van fontos szerepe, mert ezek katalizálják a DNS átrendeződését. Másrészt, az alternatív splicing mintájára, számos nanokromoszómának létezik alternatívan fragmentált formája.
(A legelső kép a innen származik.)
Swart EC, Bracht JR, Magrini V, Minx P, Chen X, et al. (2013) The Oxytricha trifallax Macronuclear Genome: A Complex Eukaryotic Genome with 16,000 Tiny Chromosomes. PLoS Biol 11(1): e1001473. doi:10.1371/journal.pbio.1001473
Bracht JR, Fang W, Goldman AD, Dolzhenko E, Stein EM, Landweber LF (2013) Genomes on the edge: programmed genome instability in ciliates. Cell 152(3): 406-16.
Nowacki M, Vijayan V, Zhou Y, Schotanus K, Doak TG and Landweber LF (2008) RNA-mediated epigenetic programming of a genome-rearrangement pathway Nature 451: 153-15.