Manapság már senki sem kapja fel a fejét, ha egy újonnan megszekvenált genom kerül szóba, a második generációs szekvenáló módszerek segítségével viszonylag olcsón ledarálható egy gerinces genom is. Időnként azonban előkerül egy-egy ötlet, amin az ember tényleg meglepődik, ilyen a ma boncolgatott cikké is: A tasmán ördög arcdaganat genomjának a bázissorrendjét határozták meg. Maga a kórokozó (Devil facial tumor disease - DFTD) egy daganatsejtvonal, ami az eredeti gazdaegyed testén kívül is életképes, tulajdonképpen fertőzőképes rák, amit harapással adnak át egymásnak a tasmán ördögök vagy tizenöt éve. A fertőzött egyedekben a sejtvonal osztódni kezd és daganatokat fejleszt, amik megölik a gazdát, éppen ezért nem csak a betegség különlegessége okán került a figyelem középpontjába, hanem azért is, mert komolyan veszélyezteti a tasmán ördög fennmaradását, valamint mivel jelenleg nem tudjuk, hogyan kerüli el az immunválaszt ez a sejtvonal, hiszen egy másik egyed sejtjeit az immunrendszernek egyszerűen el kellene pusztítania, ez a sejtvonal viszont vígan szaporodik egy csomó tasmán ördögben.
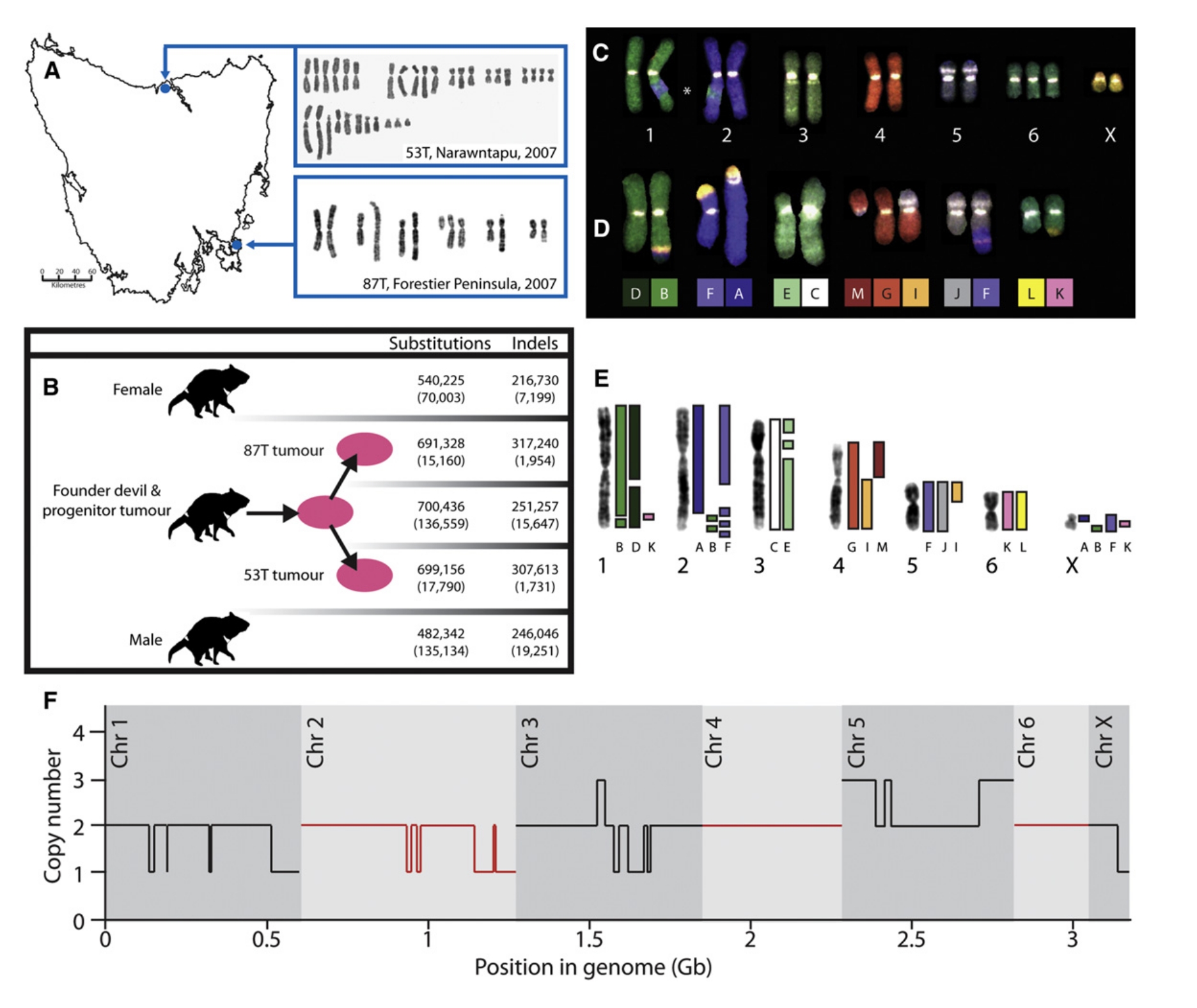
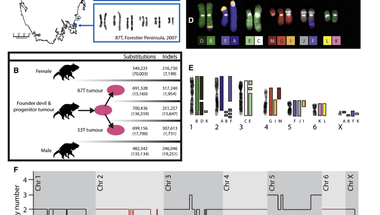
A betegséget először 1996 -ban figyelték meg, azóta egész tasmániában elterjedt, ennyi idő alatt minden bizonnyal elég sok genetikai változás történt benne az eredeti genomhoz képest. Hogy kideríthessék, mik ezek a változások, megszekvenálták egy egészséges hím és egy nőstény tasmán ördög teljes genomját és két DFTD genomot is, amelyeket egymástól távol eső vidéken gyűjtöttek be. Megállapították, hogy a tasmán ördög genomja kb. három gigabázis, az emberi genoméhoz hasonló méretű és 18775 fehérjekódoló gént tartalmaz. Ezek után összehasonlították a 87T, délkeleti DFTD sejtvonal genomjával és az 53T sejtvonal genomjával, ami tasmánia északi partvidékéről származik. Meglepő módon a két sejtvonal kariotípusa erősen különbözött egymástól és az egészséges genomoktól is, a 87T alig tér el a diploid kromoszómaszámtól, 13 kromoszómával rendelkezik, ám az 53T inkább tetraploidnak számít, 32 kromoszómával, ezek a kariotípusok láthatóak az első A ábrán. A B ábra foglalja össze a különbségeket a vizsgált genomok között, az egyes számok azoknak a változatoknak a mennyiségét mutatják, amik csak abban a genomban fordulnak elő, míg a C ábra a referenciagenomként használt nőstény tasmán ördögből származó sejtvonal kariotípusát mutatja, meglepő módon a hatodik kromoszómából kettő helyett hármat is tartalmaz. A D ábra mutatja a 87T sejtvonal kariotípusát, ebből annyi látható, hogy ivari kromoszómákat egyáltalán nem tartalmaz ellenben a negyedik kromoszómájából hárommal is rendelkezik. Az első, második és harmadik kromoszómán is jelentős méretű kromoszómarégiók hiányoznak. Az E festés hozzávetőlegesen megmutatja, milyen kromoszóma-átrendeződések történtek, a 87T DFTD sejtvonal kromoszómáit illesztették az egészséges tasmán ördög kromoszómákra, látható, hogy elég kusza a kép, az egyes kromoszómarégiók eléggé összekeveredtek ebben a sejtvonalban. Azt is megállapították, hogy az eredeti gazda valószínűleg nőstény volt, mivel az Y kromoszómára jellemző szakaszok hiányoznak a 87T genomból, ellenben két különböző X kromoszóma szakaszai mutathatóak ki a genomban. A végső összesítést az F ábra mutatja, az látható rajta, hogy az egészséges tasmán ördög genomhoz képest a 87T sejtvonal genomja mely szakaszokat milyen példányszámban tartalmazza.
Megpróbálták kikövetkeztetni, milyen mutációkat halmoztak fel az egyes DFTD sejtvonalak amióta elhagyták az első gazdaszervezetet. Abból indultak ki, hogy ami a 87T és 53T genomokban közös, az az eredeti gazdaszervezet genomja. Azt találták, hogy tizenötezer báziscsere mutációt tartalmaz ez a sejtvonal, ami nem volt meg a két DFTD sejtvonal utolsó közös ősében, az 53T pedig olyan tizenhétezer mutációt halmozott fel, amióta különvált a 87T sejtvonaltól. Ez az érték körülbelül összevethető az emberi daganatokban megfigyelhető mutációk számával.
Ez a közlemény elég jól megvilágítja a második generációs szekvenálás előnyeit és hátrányait is: Nyilván korábban elképzelhetetlen részletességgel hasonlíthatóak össze így élőlények, viszonylag olcsón. Azonban a rengeteg adatot valamilyen módon kísérletesen is érdemes ellenőrizni, ami ilyenkor nyilvánvalóan lehetetlen, senki sem fogja egyesével végigvizsgálni több százezer mutáció hatását. Két vadon élő tasmán ördög genomja közt is rengeteg eltérés figyelhető meg, nyilván a DFTD eredeti gazdaegyedének a genomja is számos eltérést hordozott mindkét referenciaegyedhez képest, nem meglepő módon a belőle származó sejtvonal is tartalmazza ezeket, de mivel az első gazda genomja nem ismert ezekről csak találgatni lehet. Az egyetlen biztos pont, hogy a két sejtvonal milyen mutációkat halmozott fel a különválásuk óta.
Elizabeth P. Murchison, Ole B. Schulz-Trieglaff, Zemin Ning, Ludmil B. Alexandrov, Markus J. Bauer, Beiyuan Fu, Matthew Hims, Zhihao Ding, Sergii Ivakhno, Caitlin Stewart, Bee Ling Ng, Wendy Wong, Bronwen Aken, Simon White, Amber Alsop, Jennifer Becq, Graham R. Bignell, R. Keira Cheetham, William Cheng, Thomas R. Connor, Anthony J. Cox, Zhi-Ping Feng, Yong Gu, Russell J. Grocock, Simon R. Harris, Irina Khrebtukova, Zoya Kingsbury, Mark Kowarsky, Alexandre Kreiss, Shujun Luo, John Marshall, David J. McBride, Lisa Murray, Anne-Maree Pearse, Keiran Raine, Isabelle Rasolonjatovo, Richard Shaw, Philip Tedder, Carolyn Tregidgo, Albert J. Vilella, David C. Wedge, Gregory M. Woods, Niall Gormley, Sean Humphray, Gary Schroth, Geoffrey Smith, Kevin Hall, Stephen M.J. Searle, Nigel P. Carter, Anthony T. Papenfuss, P. Andrew Futreal, Peter J. Campbell, Fengtang Yang, David R. Bentley, Dirk J. Evers, Michael R. Stratton (2012): Genome Sequencing and Analysis of the Tasmanian Devil and Its Transmissible Cancer. Cell Vol. 148, Issue 4, pp. 780-791