A szó etimológiája alapján azt feltételeznénk (egyébként részben helyesen), hogy a kromoszómák, vagyis "festődő testek" a sejtek talán legjobban megjeleníthető komponenseit képezik. És kétségtelen, hogy számos korabeli eljárással nagyon szépen láthatóvá lehetett tenni már egészen régen az örökítőanyag ezen nagy, diszkrét komponenseit, különösen osztódások során. Ugyanakkor a strukturális információ, ami ezen sejtkomponensekről így beszerezhető volt, viszonylag korlátozottnak bizonyult. Mégpedig azért (is), mert az a mérettartomány, ahol igazán megfigyelhető lenne, hogy miképp csomagolódik a DNS, kívül esik a fénymikroszkópok nagyítási képességein és az a helyzet, hogy a korábbi eljárásokkal a megfelelő felbontást már biztosító elektronmikroszkópokkal a kromoszómák nem festődtek.
Ez pedig azt a helyzetet eredményezte, hogy mindazt, amit a kromatin szerkezetéről, a sejtben levő DNS-lánc feltekeredéséről gondoltunk, elsősorban indirekt bizonyítékokon alapult.
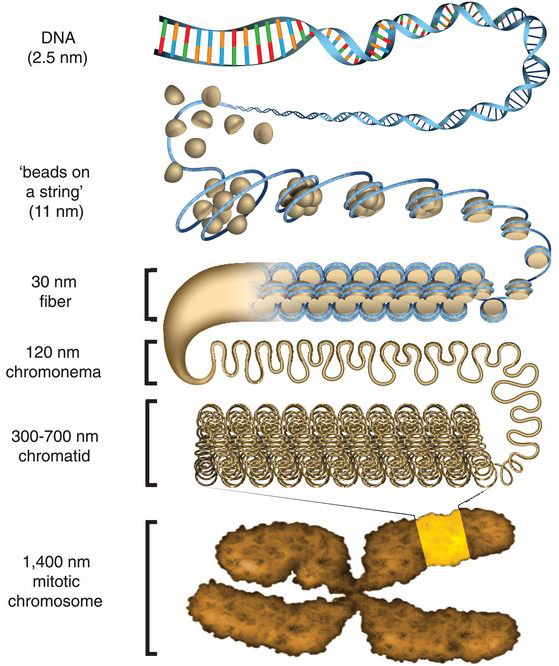 Márpedig az biztos volt a kezdetektől, hogy a közel 2 m hosszú, 10 µm átmérőjű DNS kettőslánc, ami minden egyes sejt magjában jelen van, valamilyen különleges szerveződést kell mutasson hogy elférjen és működőképes maradjon (azaz a megfelelő régiókból történjen átírás, a többiekről pedig nem).
Márpedig az biztos volt a kezdetektől, hogy a közel 2 m hosszú, 10 µm átmérőjű DNS kettőslánc, ami minden egyes sejt magjában jelen van, valamilyen különleges szerveződést kell mutasson hogy elférjen és működőképes maradjon (azaz a megfelelő régiókból történjen átírás, a többiekről pedig nem).
Ennek a szerveződésnek az első szintje, hogy a DNS ún. nukleoszómákat hoz létre a hisztonfehérjék által képzett komplex köré csavarodva (egy-egy nyolc hisztonmagfehérjéből álló komplex kb. 146 bp DNS-t tud maga köré tekerni), egy gyöngyfüzérre emlékeztető szerkezetet hozva létre.
Ha elővesszünk bármely aktuális tankönyvet, vagy jegyzetet, akkor azt is olvashatjuk, hogy a sejtosztódásban levő sejtmagban (ahol a DNS különösen tömör állapotban van), ezt a gyöngyfüzér-szerkezetet egyéb, magasabb szintű szerkezeti elemek követik: a szolenoid (30 nm átmérővel), szupercoil, majd végül a kromatidák.
A szépséghiba, hogy utóbbi szerkezeti elemeket in vitro kísérletek segítségével írták le, eddig senki nem látta őket, épp ezért tk. soha nem rendelkeztünk százszázalékos bizonyítékkal, hogy léteznek.
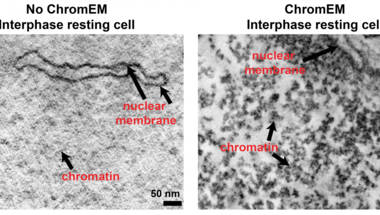
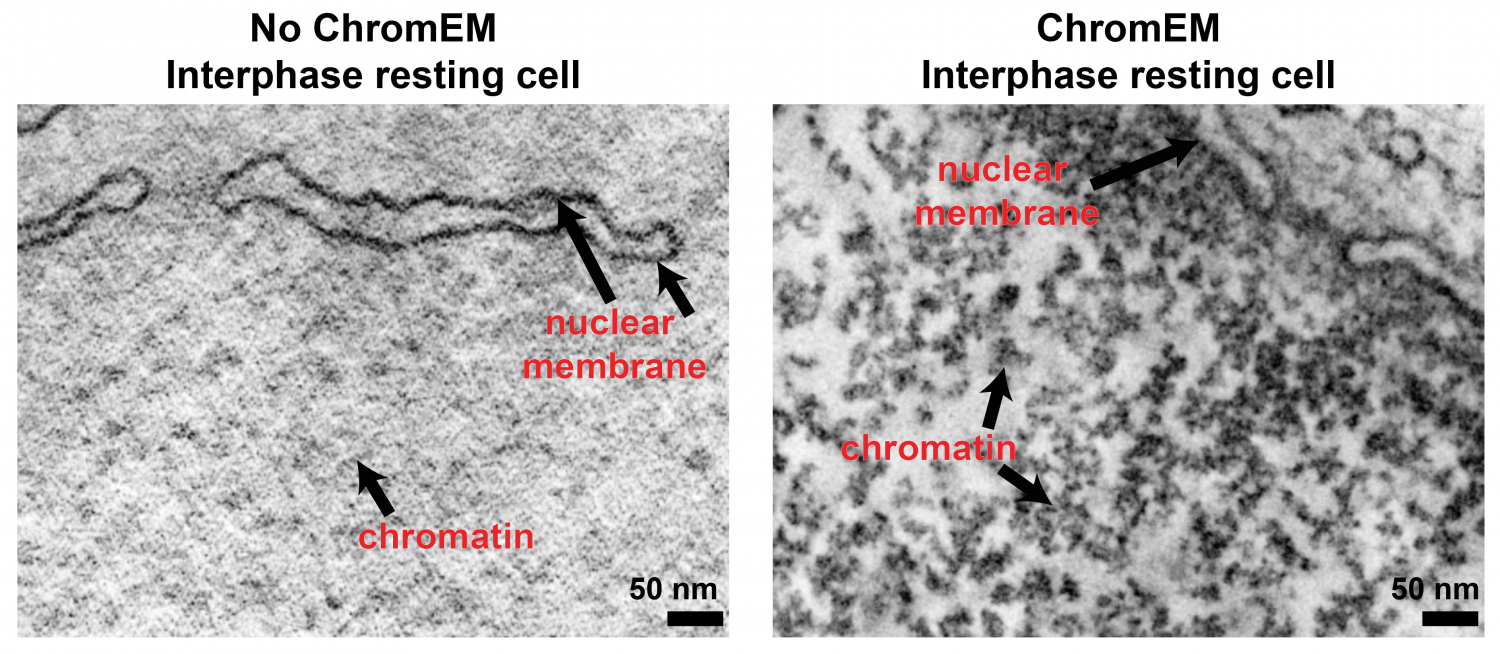
Most egy különleges DNS festék (DRAQ5) segítségével azonban lehetőség nyílt, hogy elektronmikroszkópokkal is szemügyre vegyük a sejtekben in situ is a DNS szerveződését. És mivel az EM-technológia már egészen jól kidolgozta, hogy egymás utáni metszetekből 3D-s képet miképp lehet előállítani, egyből teljes sejtmagokat tudtak kvázi rekonstruálni, mind osztódó, mind differenciált sejtekben (együttesen ezt nevezik a szerzők ChromEMT technológiának).
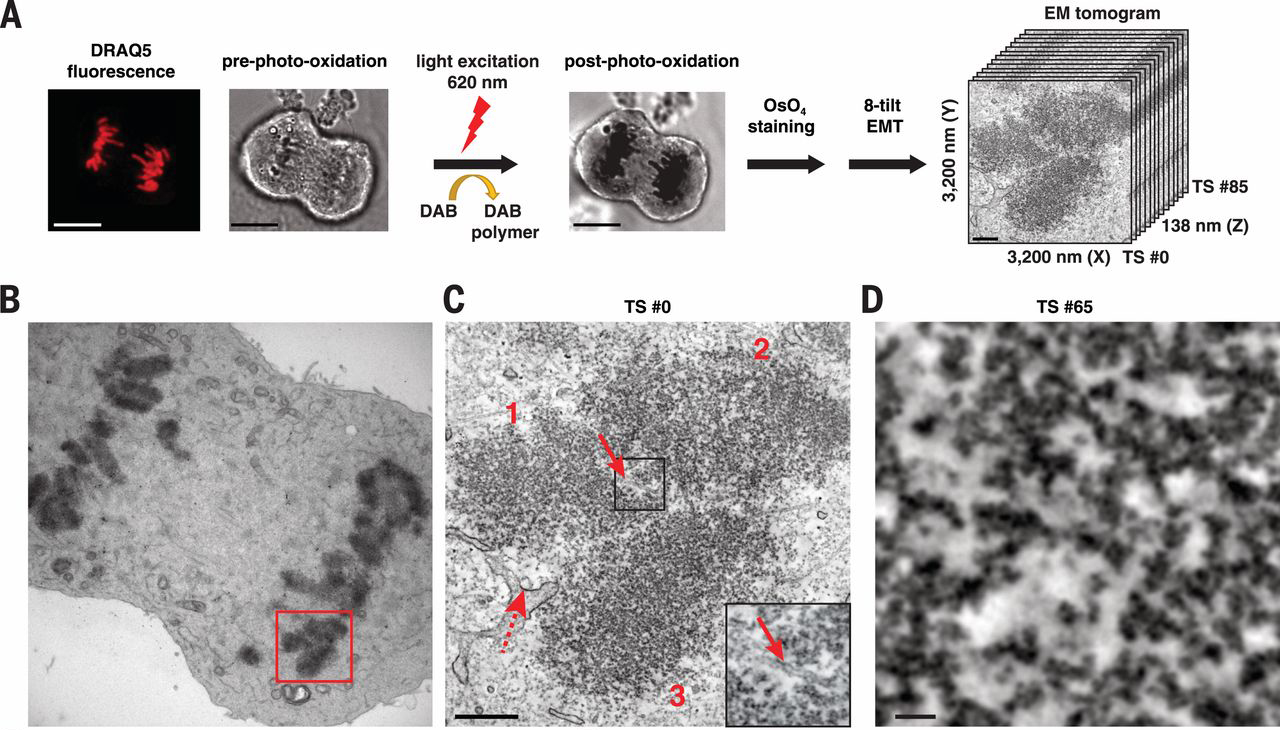
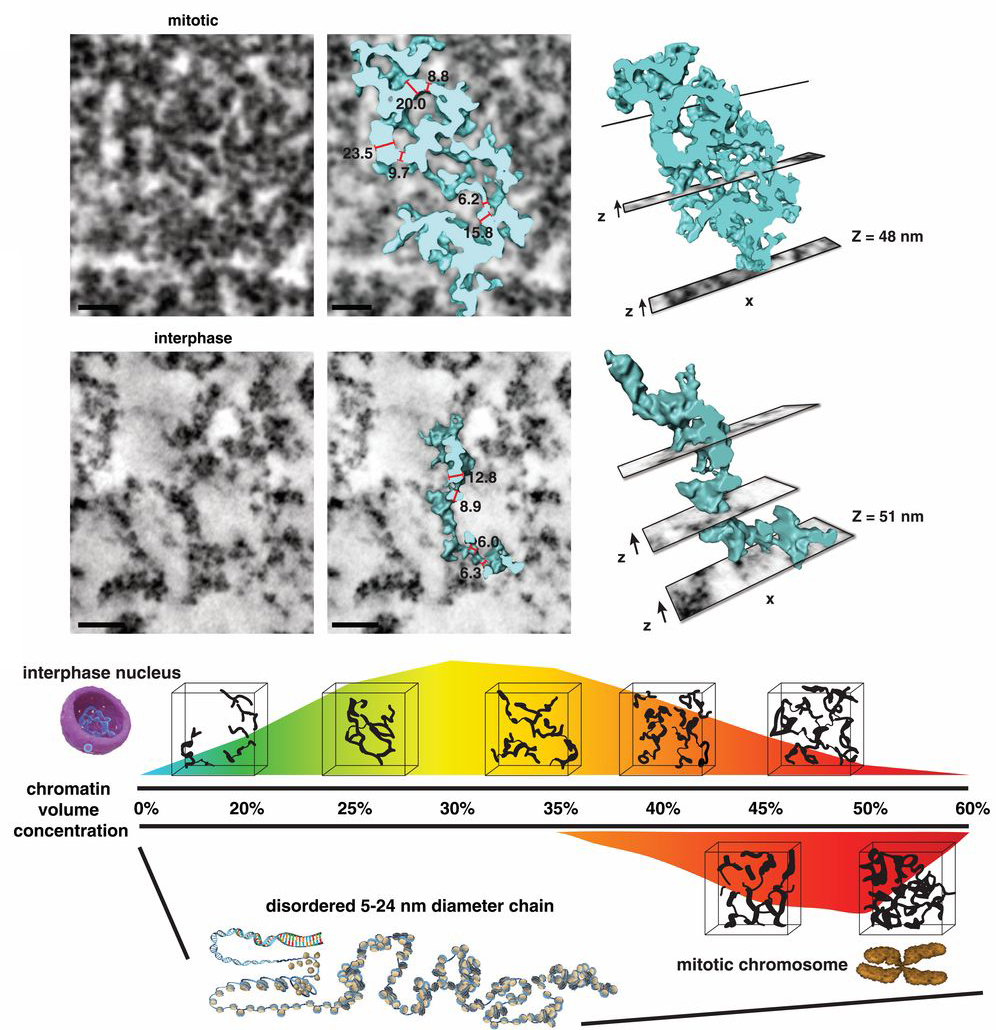
És mindenki számára talán némileg meglepő módon a korábban prediktált szerveződési hierarchiának nyoma sincs. A 3D-s rekonstrukciókban a "gyöngyfüzér" még látszik, annak rendje s módja szerint, de szolenoidot és egyébb struktúrákat nemigen lehet ezeken a felvételeken látni. A mitotikus kromoszómákban a kormatin persze tényleg tömörebb, a DNS szálak jobban egymás mellé vannak paszírozva, de összességében szervezett tömörítésnek nem lelték nyomát a szóbanforgó Science cikkben.
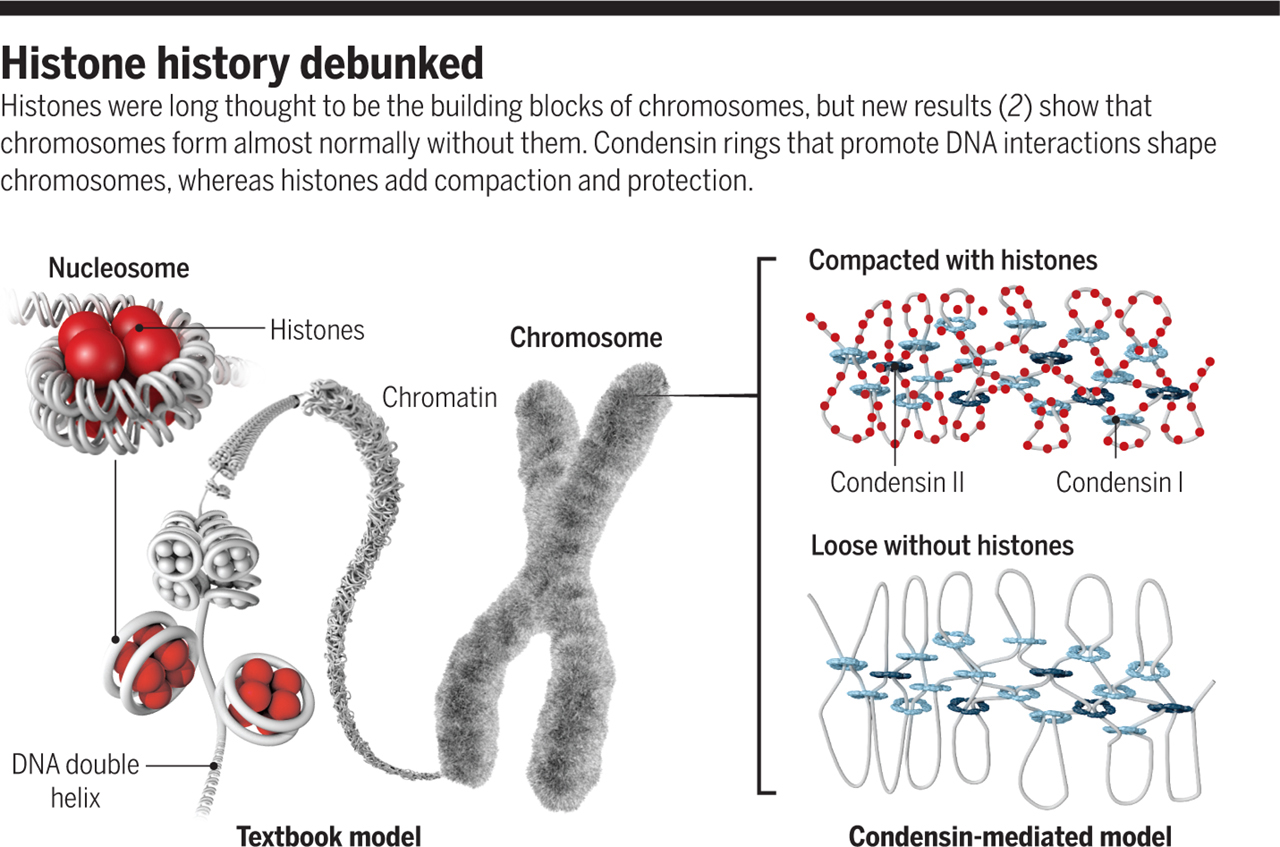
Ha pedig már kromoszóma-szerkezettel kapcsolatos tabudöntögetésnél tartunk, akkor gyorsan elővennék egy picit korábbi cikket is, amiben az említett "gyöngyfüzér" szerkezetét is egy kicsit jobban megpiszkálták.
Ebben a (szintén) Science cikkben szintetikus módon olyan körülményeket hoztak létre, amikor indukálódik a kromatin kompakttá válása: béka petesejt-extraktumba (amiből a béka DNS-t eltávolították) egér spermiumokat vittek be. Az egér spermiumban alapól nincsenek hisztonok, a DNS szerveződését protamin nevű fehérjék biztosítják, viszont a petesejt-extraktum biztosította környezet hatására a spermium DNS-e tömör kromatin szálakká ugrik össze.
So far, so good, ahogy az angolok mondanák, hiszen semmi meglepően új nincs a dologban. Csakhogy, amikor a petesejt-extraktumokból a hisztonokat és/vagy a protamin-hiszton cserét biztosító Asf1 fehérjét eltávolították, kromatin jellegű struktúrák akkor is létrejöttek. Vagyis olyan DNS is képes volt egy tömör formátumot felvenni, ami még a "gyöngyfüzér" szerkezetet sem tudta felvenni. Hogy lehet ez? A válasz elég prózai: ebben a kontextusban a hisztonokkal ilyenkor valamennyire redundáns funkciót ellátó kondenzin fehérjék játszanak fontos szerepet a DNS szálak összekötésében, és ezek a hisztonok hiányában is képesek ellátni a feladatukat - igaz, kicsit lazább szerkezet jön létre.
Ha pedig már egyre inkább átfolyik ez a poszt kötetlen, kromoszóma-centrikus egyveleggé, még álljon itt távirati rövidséggel két másik érdekesség is, az elmúlt hónapok terméséből, ami szintén hozzá tesz a tankönyvek klasszikus kromoszóma képéhez.
Persze egy rövid be- és átvezetőt aligha lehet megúszni, mert azért a két cikk amiről nagyon röviden említést tennék, azért vastagon épít az utóbbi kb. 5-6 év tudásrobbanárására, ami a kromoszómák sejtmagban való elhelyezkedésének témájában történt. Ez esetben nem kifejezettem osztódó sejtek, hanem a sejtciklusból kilépett, aktív transzkripciót folytató sejtekre is koncentráltak, hiszen azt is érdekes feltérképezni, hogy miképp néz ki a kromoszómák szerkezete az ilyen "hétköznapi" állapotok során. Mindehhez szükség volt egy metodológiai ugrásra is, egyrészt szekvenálás, másrészt számítókapacitás terén, mert ezek a módszerek arra építenek, hogy adott pillanatban megkísérlik feltértképezni, egy-egy kromoszómaszakasz milyen más szakaszok közelében helyezkedik el (ne adj ég, érintkezik), és ezekből az interakciókból hogyan lehet egy 3D-s sejtmagtérképet visszakapni.
Ami világosan látszik, hogy az egyes kromoszómák a sejtmagon belül jól meghatározott territóriumokat töltenek ki (chromosome territories), és az is megfigyelhető, hogy a kromoszómán belüli kapcsolatok nagyságrenndel nagyobbak, mint a kromoszómák közöttiek. A kromoszóma territóriumon belül transzkripcionálisan aktív A és inaktív B kompartmenteket lehet elkülöníteni (a klasszikus eukromatin és heterokromatin körülbelüli megfelelői), és ha már kromoszómák közti kapcsolatokat nézünk akkor az azonos típusú kompartmentek közti kapcsolatok gyakoribbak, mint az eltérő kompartmentumok közöttiek, és az A-A típusú kapcsolatok összességében még gyakoribbak, mint a B-B kölcsönhatások.
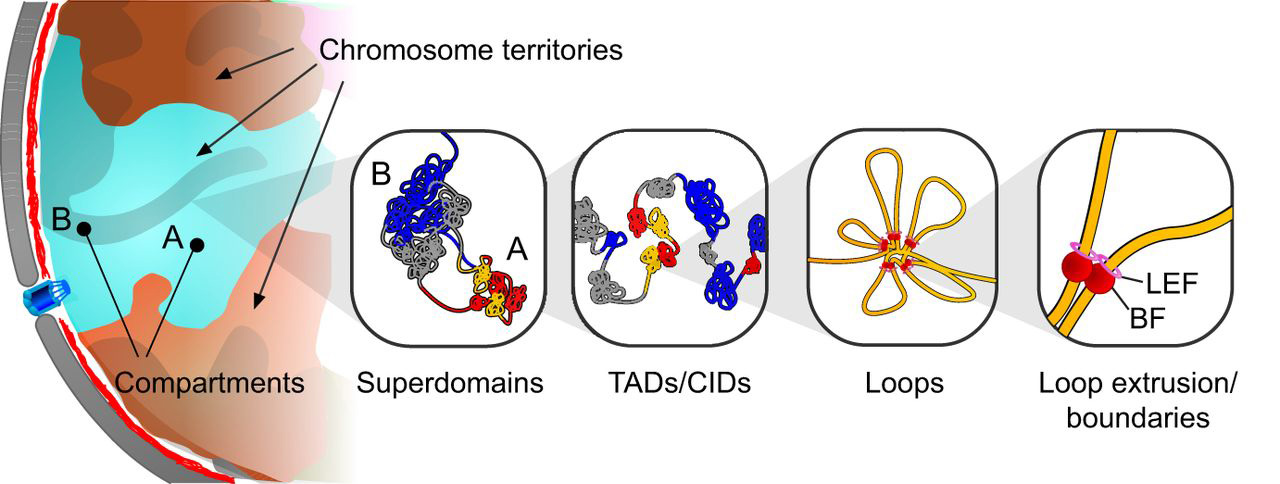
Ha tovább közelítünk, akkor eljutunk az ún. TAD-ok (topologically associated domains) világába, amelyek olyan DNS doméneket jelölnek, ahol már igazán gyakoriak az interakciók - gyakran ez azzal jár együtt, hogy egy gén és aktív enhanszerei kerülnek egyetlen TAD-ba, és az ezek közötti interakciók további hurkok kialakulásával járnak. Összességében még talán azt fontos megjegyezni, hogy a lokálisabb kapcsolatok a kiszámíthatóbbak és reprodukálhatóbbak különböző minták között, a globálisabbak már sokkal kevésbé.
Itt tartottunk kb. év elején és két elegáns, a Nature-ben megjelent tanulmány tovább finomított a képen. Az elsőben (igazi technológiai tour de force-ként) egy-egy sejt esetében sikerült a kromoszómaterületek és A/B kompartmentek szerkezetét feltárni (és összevetni, hogy miképp viszonyul az egyes sejtek sejtmagszerkezete egymáshoz).
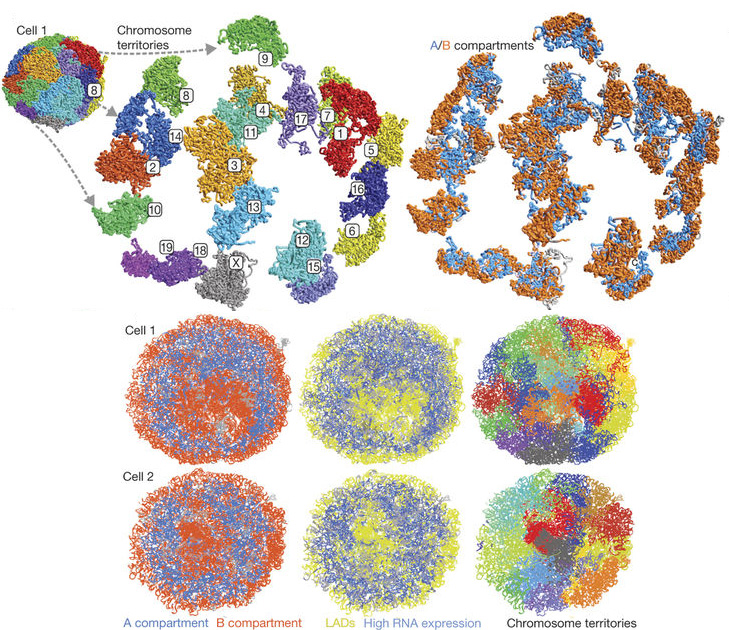
Ez pedig megerősítette, hogy az A kompartment minden kromoszóma esetében inkább a sejtmag belsejében található és aktív transzkripció jelöli, míg a sejtmagmembrán felépítésében résztvevő laminokkal kapcsolatban levő lamin-asszociált domének (LAD) a transzkripcionálisan inaktív B kompartmentnek felelnek meg. Ugyan a kormoszómák alakja különböző sejtmagokban nagyon eltérő lehet és a sejtmag más-más relatív pontjában találhatjuk őket, az is jól látszik, hogy a B kompartmentbe kerülő szekvenciák, azok mindig ugyanoda kerülnek, míg az A-ba tartozók pedig mindig az aktív transzkripciós területre (azonos típusú sejtekben).
Az utolsó cikk pedig ugyanezeket az egysejt-technológiákat a sejtciklus különböző fázisaiban levő sejtmagokra alkalmazta. A legérdekesebb, ami ebből a kutatásból visszaköszönt, hogy az A és B kompartmentumok legjobban nem is annyira a sejtcikluson kívül, vagy annak az elején, hanem közvetlenül a mitózis előtt, a G2 fázisban detektálhatók legjobban - hogy miért, az még nem teljesen világos, de mindenképpen érdekes megfigyelés.
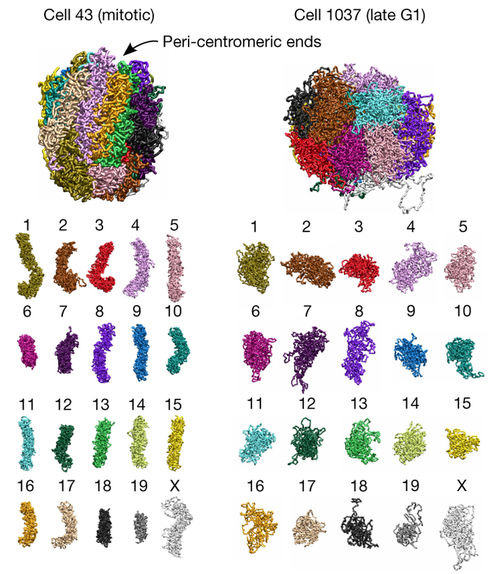
Na és persze, az is szépen látszik, hogy miképp tömörödnek be a kromoszómák a mitózis előtt, globuláris, laza szerkezetből átváltanak tömör, rúdszerű struktúrába - ami persze egyáltalán nem meglepő, hiszen már a klasszikus festési eljárások is ezt mutatták, de itt egy festéstől független módon is láthatjuk, ami viszont jól igazolja ezeknek a konformáció-detektáláson alapuló eljárásoknak a pontosságát és létjogosultságát.
Larson DR, Misteli T (2017) The genome-seeing it clearly now. Science 357(6349): 354-355.
Ou HD, Phan S, Deerinck TJ, Thor A, Ellisman MH, O'Shea CC (2017) ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science 357(6349) pii: eaag0025.
Kakui Y, Uhlmann F. (2017) Building chromosomes without bricks. Science 356(6344): 1233-1234.
Shintomi K, Inoue F, Watanabe H, Ohsumi K, Ohsugi M, Hirano T (2017) Mitotic chromosome assembly despite nucleosome depletion in Xenopus egg extracts. Science 356(6344): 1284-1287.
Nagano T, Lubling Y, Várnai C, Dudley C, Leung W, et al. (2017) Cell-cycle dynamics of chromosomal organization at single-cell resolution. Nature 547(7661): 61-67.
O'Shaughnessy-Kirwan A, Cramard J, Faure AJ, Ralser M, Blanco E, et al. (2017) 3D structures of individual mammalian genomes studied by single-cell Hi-C. Nature 544(7648): 59-64.
Pueschel R, Coraggio F, Meister P (2016) From single genes to entire genomes: the search for a function of nuclear organization. Development 143(6): 910-23.