 Kevés ma már nem élő állat képes annyi embert izgalomba hozni, mint a mamut. És valahogy az első befagyott mamutok felfedezése óta tartja magát a remény/vágy, hogy egyszer fel tudjuk majd támasztani ezeket a különleges, szőrös elefántokat.
Kevés ma már nem élő állat képes annyi embert izgalomba hozni, mint a mamut. És valahogy az első befagyott mamutok felfedezése óta tartja magát a remény/vágy, hogy egyszer fel tudjuk majd támasztani ezeket a különleges, szőrös elefántokat.
Fél éve írtam ezekről a törekvésekről, illetve arról, hogy ezek közül is a legígéretesebb nem is annyira mamut-klónozással érne fel, mint egyszerűen az ázsiai elefánt "mamutosításával". (Közben a kolléga is írt erről röviden a ScienceMeetup blogján.)
Utóbbi azt jelentené, zanzásítva, hogy a mamut-genom ismeretében meg tudjuk mondani, hogy hol vannak azok a mutációk, amelyek megkülönböztették ezeket az egykor élt lényeket mai rokonaiktól, és ezeket a változásokat aztán bevinnénk az elefánt genomba; a sejteket felhasználnánk egy kiméra-elefánt létrehozására, és ha annak az ivarvonalába bekerülnek, akkor a következő generációban már részben "mamutosított" elefántok születnének.
Ehhez persze elengedhetetlen, hogy ne egyetlen mamut-genom álljon a rendelkezésünkre, hiszen akkor hogy is tudnánk eldönteni, hogy egy-egy hely, ahol ez különbözik az ázsiai elefántok genomjától egyszerű, nem sok jelentéssel bíró polimorfizmus, vagy valami nagyon is fontos, faj-specifikus mutációt kódoló DNS szakasz?
Épp ezért az utóbbi időben egyre több mamut genomja került megszekvenálásra: az elmúlt hónapokban éppen négy, amelyek közül kettőt a Current Biology-ban publikáltak, kettőt pedig egy, egyelőre csak a bioRxiv felületén elérhető cikkben.
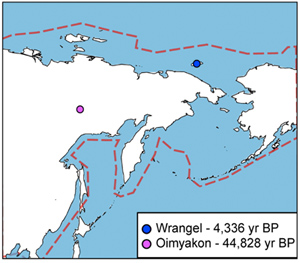 Az előbbi esetben a két minta térben és időben igencsak eltérő populációkból származott: az egyik mamut a jakutföldi Ojmjakon környékéről került elő és majdnem 45 ezer évvel ezelőtt élt, a másik pedig már, a mamutok alkonyán, ~4300 évvel ezelőtt, a Vrangel-szigeten.
Az előbbi esetben a két minta térben és időben igencsak eltérő populációkból származott: az egyik mamut a jakutföldi Ojmjakon környékéről került elő és majdnem 45 ezer évvel ezelőtt élt, a másik pedig már, a mamutok alkonyán, ~4300 évvel ezelőtt, a Vrangel-szigeten.
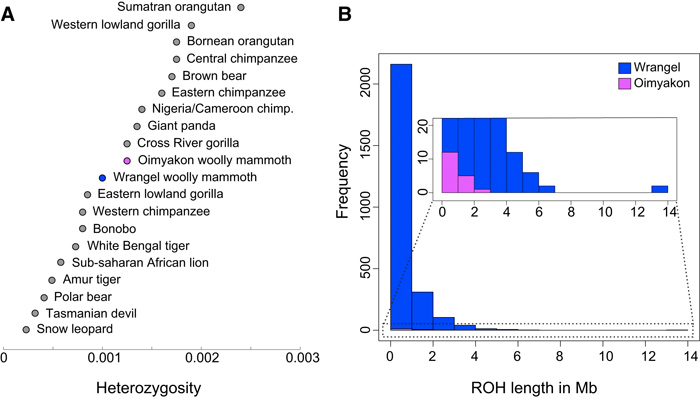
A két egyed összevetése így egyrészt informatív azért is, hogy lássuk, mennyit változott ennyi idő alatt a mamutok genomja, de ami még ennél is fontosabb: abból, hogy ezek a diploid genomok mennyi polimorfizmust hordoznak (vagyis hány olyan helye van a genomnak, ahol a kromoszómapárok megfelelő pozíciójában eltérő szekvencia van, azaz az egyed heterozigóta arra a szakaszra nézve), arra is következtethetünk, hogy mennyire volt beltenyésztett az adott helyen és időben élő mamutcsorda.
Az eredmények, mint az alábbi ábra is mutatja, talán nem is túl meglepően arra engednek következtetni, hogy a heterozigócia fokozatosan lecsökkent (az ROH érték azt mutatja, milyen hosszú szakaszok kizárólag homozigóták, azaz tökéletesen azonosak, a genomban) a vrangel-szigeteki egyedekben (kb. 20%-al az ojmjakoni egyedhez képest), ami jól jelzi, hogy ezek az utolsó mohikánok már csak egy kihalófélben levő populációt képviseltek. (Ugyanakkor - és ez szomorú a jelenre nézve - az ő heterozigócia szintjük még mindig magassabb volt, mint számos ma veszélyeztetett fajé...)
 A másik tanulmány kevésbé foglalkozott populációgenetikával, őket sokkal jobban érdekelte, hogy _általában_ milyen génekben különbözik a mamut és ázsiai elefánt genom, illetve, ha valamilyen érdekes génben találnak mutációt, fel tudják-e "támasztani" azt a gént.
A másik tanulmány kevésbé foglalkozott populációgenetikával, őket sokkal jobban érdekelte, hogy _általában_ milyen génekben különbözik a mamut és ázsiai elefánt genom, illetve, ha valamilyen érdekes génben találnak mutációt, fel tudják-e "támasztani" azt a gént.
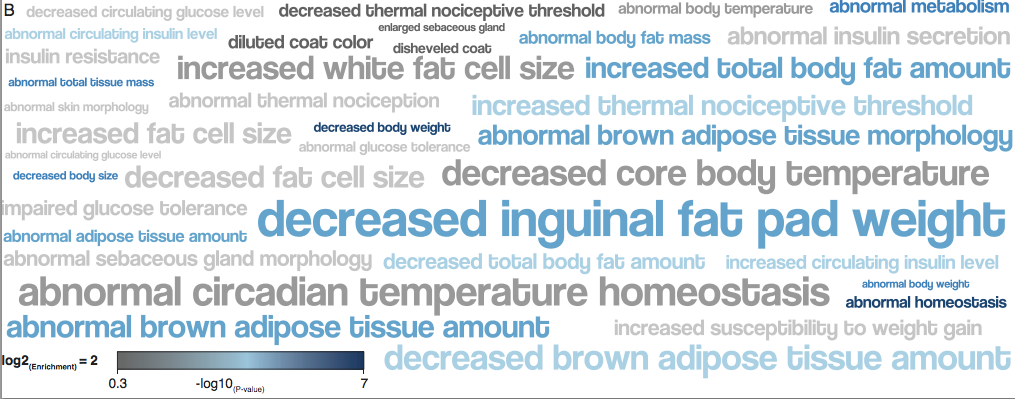
A genomok-összevetése után összegyűjtötték azokat a fehérjekódoló géneket, amelyekben valami jellegzetes aminosav-csere történt és egy másik adatbázisban megnézték, hogy ezeknek a géneknek milyen szerepük van a klasszikus emlős-model, az egér esetében. Az eredmény leginkább azért hihető, mert nem meglepő: az alább is látható szó-felhő egyértelműen a zsír anyagcserében bekövetkezett változásokat emeli ki, ami várható, hiszen a hideg éghajlathoz a mamutok a zsírfelhalmozással (is) alkalmazkodtak.
Ehhez kapcsolódik az RNASEL (Ribonuclease L) gén kópiaszámában bekövetkezett változás is (elefántban minden sejtben 2, mamutban 6 db), hiszen ez is a zsírsejtek differenciációját szabályozza.
Kicsit más tészta a később "felélesztett" TRPV3 gén, ami a hőérzékelést szabályozza (a magas, 33°C feletti hőmérsékletet jelzi), illetve a szőrnövekedésben van fontos szerepe. A gén mamut-verziója olyan mutációt hordoz (N647D), ami a laboratóriumi körülmények között (egy humán, vesesejtekből álló sejtvonalba, a HEK293-ba vitték be) kevésbé aktív, mint az elefánt megfelelője. Ez pedig nagyon emlékeztet arra, amit Trpv3-mutáns egerekben megfigyelhetünk. És ezért is adja magát, hogy utóbbiak egy másik fenotípusát is figyelmbe vegyük (amit a HEK293 sejtekben nem lehetett letesztelni): amikor a TRPV3 nem működik rendesen (vagy egyáltalán), az egerek szőre hosszabb és hullámosabb lesz - pont mint a mamutoké. Vagyis a TRPV3(N647D) pont egy olyan mutációnak tűnik, amit az elefántok mamutosításához hasznosíthatnánk.
Persze a nagy kérdés továbbra is az marad, hogy van e létjogosultsága az egész mamut-deextinkciós projektnek. A korábbi posztban már írtam George Church támogató álláspontjáról, most egy másik fajsúlyos személy, Beth Shapiro szekptikusabb véleményéről ejtenék pár szót, főleg mert a hölgynek most jelent meg How to Clone a Mammoth címmel a nagyon is témábavágó műve (itt lehet olvasni Carl Zimmer kritikáját a könyvről). Shapiro szerint - és a véleményét nemrég a ScieneFriday adásában is kifejtette, ezt hallgathatjátok meg alább - egyáltalán nem triviális, hogy minden, deextinkciós program alanyaként szereplő állatot valóban megéri visszahozni. Nagyon fontos lenne azt felmérni, hogy egyáltalán létezik-e még az a niche, amit az adott faj valaha betöltött, valóban haszna származik-e az ökoszisztémának abból, ha újból megjelenik-e ez a faj, és mennyiben realisztikus azt feltételezni, hogy egy teljesen mesterséges körülmények között létrehozott hibrid képes lesz a korabeli rokonai viselkedésmintáit produkálni. (Shapiro például különösen szkeptikus a vándorgalambok deextinkciójával kapcsolatban, pedig azok genomját pont az ő laborjában szekvenálják mostanság.)
(A kezdőkép a Natural History Museum oldaláról származik.)
Palkopoulou E, Mallick S, Skoglund P, Enk J, Rohland N, et al. (2015) Complete genomes reveal signatures of demographic and genetic declines in the woolly mammoth. Curr Biol 25(10): 1395-400.
Lynch V, Bedoya-Reina OC, Ratan A, Sulak M, Drautz-Moses DI, et al. (2015) Elephantid genomes reveal the molecular bases of Woolly Mammoth adaptations to the arctic. Preprint at bioRxiv doi: 10.1101/018366.







