Az már-már közmondásos, hogy a genomok tele vannak mindenféle evolúciós hordalékkal, de hogy mégis hogyan kell ezeket elképzelni, arra szeretnék ma egy példát mutatni. Ugráló genetikai elemekről már volt ezen a blogon, például a P-elemről, de ma egy gerinces példát szeretnék mutatni.
Az már-már közmondásos, hogy a genomok tele vannak mindenféle evolúciós hordalékkal, de hogy mégis hogyan kell ezeket elképzelni, arra szeretnék ma egy példát mutatni. Ugráló genetikai elemekről már volt ezen a blogon, például a P-elemről, de ma egy gerinces példát szeretnék mutatni.
 Az már-már közmondásos, hogy a genomok tele vannak mindenféle evolúciós hordalékkal, de hogy mégis hogyan kell ezeket elképzelni, arra szeretnék ma egy példát mutatni. Ugráló genetikai elemekről már volt ezen a blogon, például a P-elemről, de ma egy gerinces példát szeretnék mutatni.
Az már-már közmondásos, hogy a genomok tele vannak mindenféle evolúciós hordalékkal, de hogy mégis hogyan kell ezeket elképzelni, arra szeretnék ma egy példát mutatni. Ugráló genetikai elemekről már volt ezen a blogon, például a P-elemről, de ma egy gerinces példát szeretnék mutatni.
Ugye a P-elemet úgy fedezték fel, hogy a helyváltoztatása nyomán keletkező mutációkat figyelték meg. Ezzel szemben a csipkerózsika ugráló genetikai elem története éppen fordítva kezdődött, Ivics Zoltán és munkatársai a lazac genomban felfigyeltek egy nagy kópiaszámban meglévő ugráló genetikai elemre, azonban a DNS szekvenciájuk alapján azonosított transzpozonok annyi különböző mutációt hordoztak, hogy egyikük sem működött, képtelenek voltak helyet változtatni a genomban. Mivel sok példányt találtak és ezeket sokféle különböző mutáció tette működésképtelenné, számos ugráló genetikai elem összehasonlításából ki tudták következtetni a működőképes csipkerózsika elem DNS szekvenciáját. Egészen pontosan nyolc különböző halfajban talált tizenkét ugráló genetikai elemet hasonlítottak össze, így állították elő a csipkerózsika konszenzusszekvenciáját, ez látható az első ábrán.
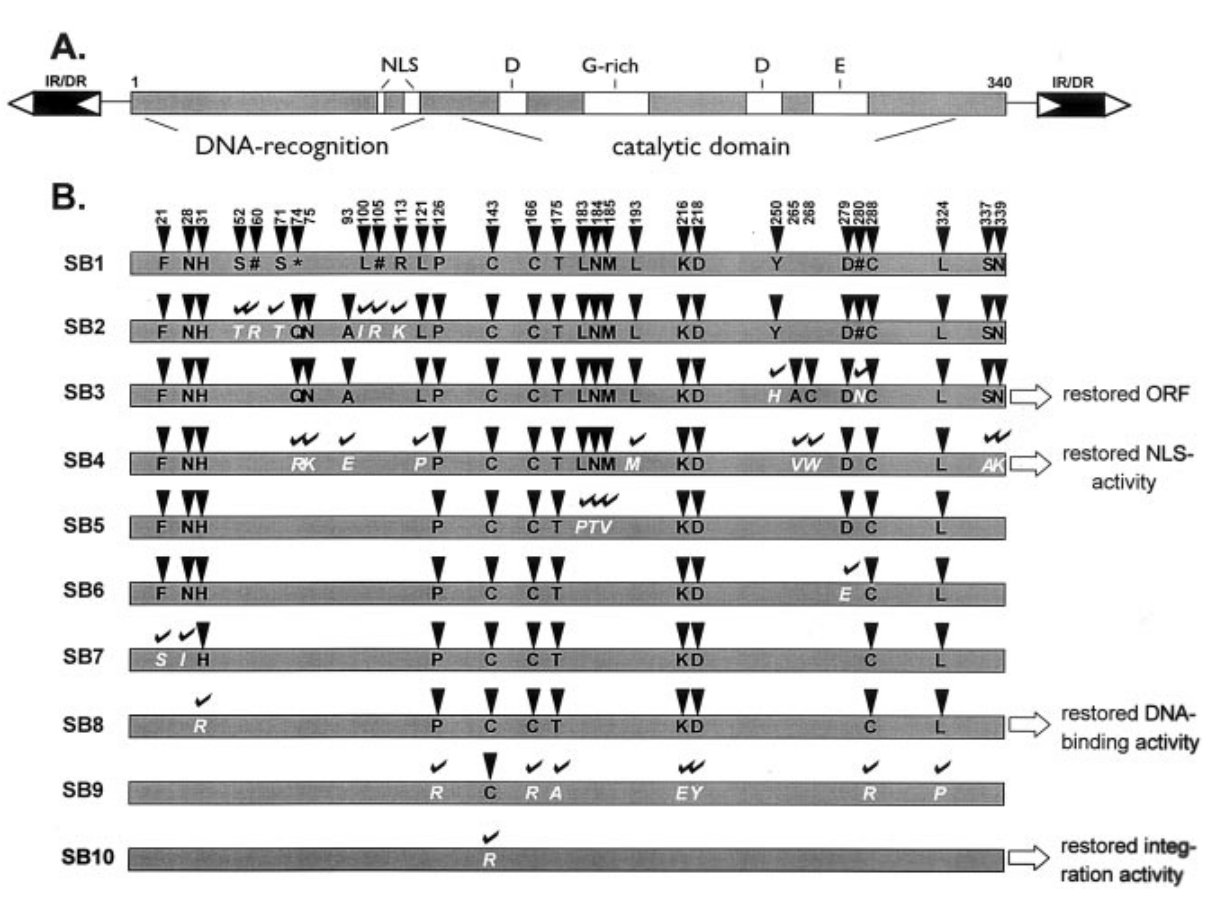
Viszont nem álltak meg itt, két lazac eredetű és egy szivárványos pisztráng eredetű szekvenciából a valóságban is összeállították a csipkerózsika ugráló genetikai elemet. Ez még mindig nem működött, számos mutációt tartalmazott, amik korai STOP kodonokat eredményeztek, valamint számos aminosavcserével jártak a fehérjetermékben. Ezeket a mutációkat egyesével kijavították PCR mutagenezis módszerrel (ez látszik az ábrán), míg meg nem kapták a csipkerózsika ugráló genetikai elem ősi változatát. Ez a transzpozáz enzim már képes volt kiugratni a csipkerózsika elemet a helyéről, vagyis a rekonstruált ősi pont úgy működött. Mivel több lépésen kersztül jutottak el az SB10 génhez, jó pár mutáns fehérjét kódoló gént is kaptak melléktermékként, ezekből megállapíthatták, a fehérje mely szakaszai mely működésért felelősek. Ezek után egy baktériumban megtermeltették a fehréje első 123 aminosavnyi szakaszát ami a fehérje DNS kötő doménjét tartalmazza és kipróbálták, hogy kötődik -e DNS molekulához? Nem meglepő módon azt tapasztalták, hogy ez a rekombináns fehérje kötődik az ugráló genetikai elem szignálszekvenciájához.
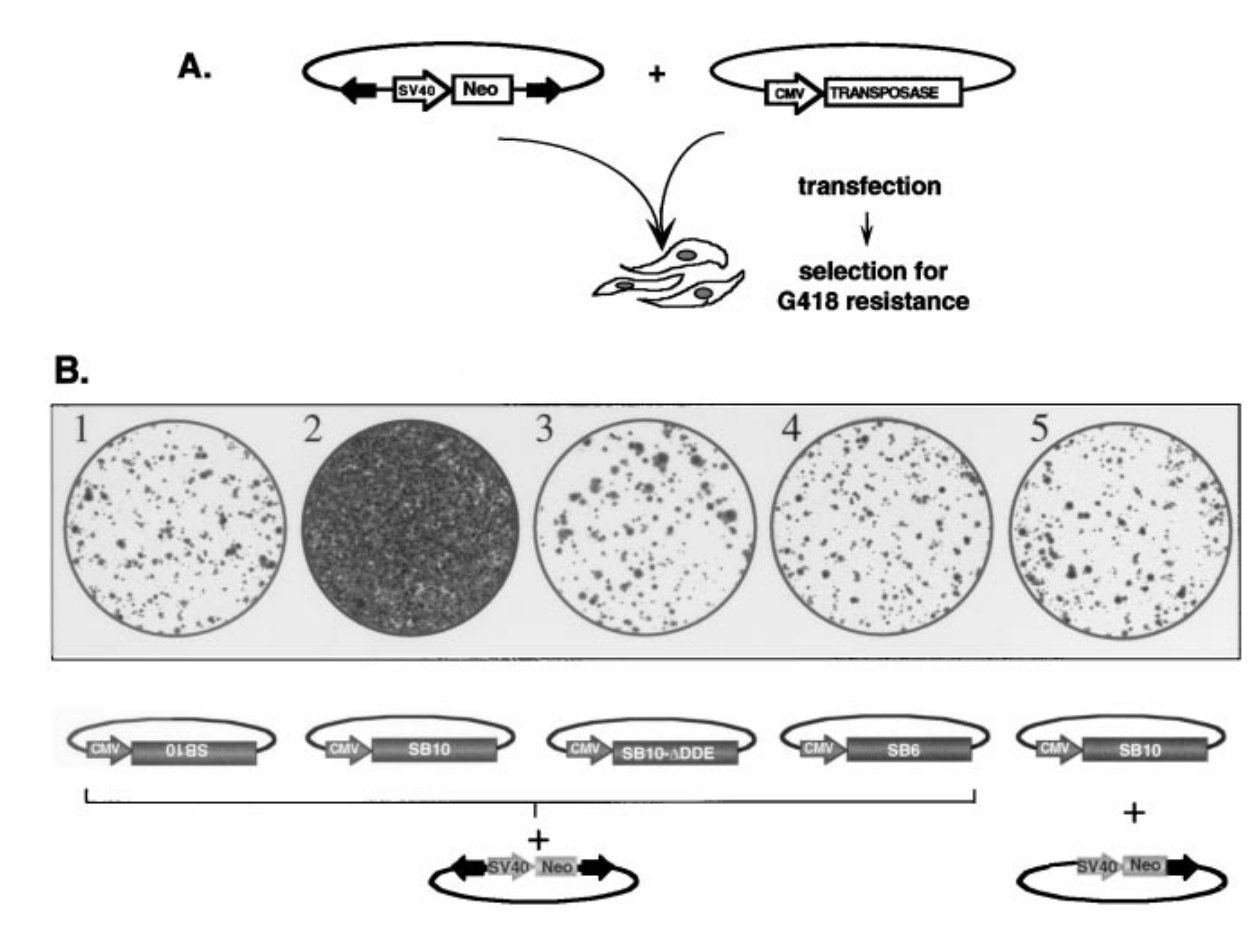
Ezek után kirpóbálták, hogy a csipkerózsika transzpozáz enzim katalizálja -e az ugráló genetikai elem beépülését egy gerinces genomba? Egy emberi sejtvonalban (HeLa) vizsgálták az SB10 fehérje működését. A transzpozáz működésére használt mérési módszer két elemű: Egy plazmidra egy neomicin rezisztancia gént klónoztak az ugráló genetikai elem szignálszekvenciái közé, ez a reporter plazmid. Ha ez beépül a genomba, akkor a sejtek egy része ellenálló lesz a neomicinre. A másik plazmid pedig az éppen vizsgált transzpozáz enzimet kódoló gént tartalmazta, az ugráló genetikai elem szignálszekvenciái nélkül, így ez nem változtatja a helyét. Az eredmény a második ábrán látható: Az első kísérletben az SB10 fehérjét kódoló gént fordított irányultsággal építették be a plazmidba, ez láthatóan nem működött. A második mintában a helyes irányultsággal építették be az SB10 fehérjét kódoló gént, ez láthatóan jelentősen megnövelte az ugráló genetikai elem beépülésének az esélyét. A harmadik kísérletben a beépített gén egy olyan SB10 fehérjét kódolt, aminek hiányzott a C-terminális végéről a katalitikus domén, ez sem volt képes a genomba építeni az ugráló genetikai elemet. A negyedik kísérletben az SB6 fehérjét vizsgálták, ez láthatóan nem működött, az ötödikben pedig egy rekombinációs szignálszekvencia nélküli ugráló genetikai elemet próbáltak ugratni az SB10 fehérjével, ez sem működött. Ellenőriztek néhány neomicin rezisztens sejtvonalat, nem meglepő módon ezekben az emberi genomban találták meg az ugráló genetikai elemet.
Összefoglalva tisztán genomszekvenciákból kiindulva azonosítottak egy ősi ugráló genetikai elemet, ami millió évek kel ezelőtt működésképtelenné vált. Hogy igazolják, ez tényleg egy egykori ugráló genetikai elem, a különböző mutációkat hordozó elemekből összeállították az ősi csipkerózsika elem szekvenciáját és el is készítették ezt a transzpozont, amely működőképesnek bizonyult egy emberi sejtvonalban is. Ha úgy tetszik kinyomozták a junk DNS egy típusának az eredetét.
Ivics Z, Hackett PB, Plasterk RH, Izsvák Z. (1997): Molecular reconstruction of Sleeping Beauty, a Tc1-like transposon from fish, and its transposition in human cells. Cell. 91(4):501-10.