Evolúcióbiológusként az első bejegyzésem itt fura módon az intelligens tervezésről szól. Lassan ott tartunk a világ ismeretében, hogy egészen érdekes dolgokat tudunk racionálisan tervezni. Persze tervezni már régóta tervezünk dolgokat, mint épületeket, járműveket vagy akár számítógépeket. De gyógyszereket? Manapság azokat is.
Olyan betegségekről legyen szó, amelyben egy RNS a ludas. Lehet ez egy RNS vírus vagy egy rák, amelyben egy szabályzó RNS molekula felelős a kontrollálatlan sejtosztódásért. Az RNS molekulák másodlagos szerkezetét (lásd ábra) könnyű számítógépes módszerekkel jósolni. Az így kapott szerkezetben levő elemek ellen kell valamilyen szelektív kis molekulás gyógyszert készíteni, ami kötődve az RNS molekulához, akadályozza annak működését.
Ez így leírva egyszerűnek hangzik, de honnan fogjuk tudni, hogy milyen vegyület kell ezen RNS-en levő szerkezeti egységek ellen? Nem árt, ha van egy adatbázis pont erre a célra. Az elmúlt években Matthew Disney és kutatócsoportja pontosan ilyen adatbázist hozott össze.
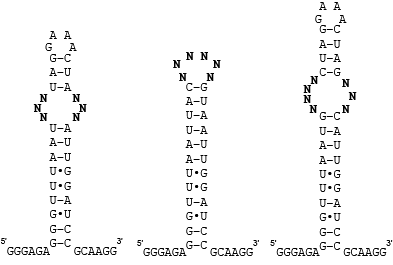
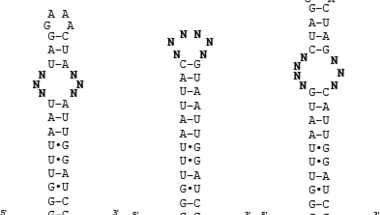
Vizsgált RNS-ek másodlagos szerkezete. A másodlagos szerkezetben azt tüntetjük fel, hogy mely bázis mely másik bázissal kapcsolódik (s persze mely bázisok vannak magukban). A legismertebb másodlagos szerkezet a tRNS "lóherelevél" formája. A félkövérrel szedett N-ek jelölik azokat pozíciókat, amelyekre mindenféle kombinációban nukleotidokat helyeztek, s azok kötődését kis molekulákhoz nézték. A jobb és baloldali RNS-ben egy belső hurkot változtattak, míg a középsőnél egy véghurkot.
Az adatbázis alapjául szolgáló adatokat az úgynevezett kétdimenziós, kombinációs szűréssel (two-dimensional combinatorial screening, 2DCS) szerezték. Röviden, vegyünk egy felszínt, amire rakosgassunk fel molekulákat. Ezek lesznek azok a kis molekulák, amelyekről szeretnénk, ha kötődnének valamilyen RNS-hez. Ezeket a molekulákat jól odarakjuk (kémiailag odakötjük, immobilizáljuk). Majd vegyünk egy RNS könyvtárat (azaz sokféle RNS molekulát), amelyben egyetlen belső hurok minden lehetséges változatát előállítottuk. Ez a 6 nukleotidból álló rész összesen 4096 (4×4×4×4×4×4) különböző belső hurkot jelenthet (fenti ábra bal oldali szerkezet). Ezeket eresszük rá az immobilizált kis molekulákra úgy, hogy erős kapcsolódás esetén az RNS molekulák is maradjanak a helyükön. Így később az RNS molekulát kinyerve az felszaporítható PCR-el és szekvenciája meghatározható (hogy tudjuk mi kötődött mihez). Persze a kis molekula nem csak ehhez a 6 nukleotidhoz vagy az általuk kialakított szerkezetekhez kötődhet, hanem az RNS molekula más részeihez is. Minket viszont csak ez a kis részlet érdekelt (ebből is nagyon sok fajta van). Legyártották az RNS-eket úgy is, hogy ez a belső hurok nem szerepel bennük. A kötődésvizsgálatot ezzel a szerkezettel is elvégezve, ha az kötődik ugyan azon kis molekulához, akkor nem a 6 bázisos részen keresztül köt. Ha viszont a belső hurok nélkül nem köti az RNS a kis molekulát, akkor bizony a minket érdeklő részlet felelős a kötődésérét.
A kis molekula kötésére szelektált szekvenciákban/szerkezetekben aztán megkeressük a hasonlóságot (például, hogy UU egymással szemben van, vagy az AC nem párosodó kitüremkedés (belső hurok) mellett legalább egy GC pár is van). Ez a számítógépes része a módszernek. Magának az RNS másodlagos szerkezetnek a meghatározása is számítógépes módszerrel történik.
Az emlőrák kialakításában feltehetőleg részt vevő microRNA-96 nevű szabályozó RNS elnyomja a FOXO1 sejthalált elősegítő fehérje kifejeződését. Így a sejtek nem halnak meg, amikor meg kéne halniuk, s kontrollálatlanul osztódni kezdenek. Ezt az RNS-t egy bis-benzimidazol köti (korábbi tanulmányukból ismerték (Velagapudi et al. 2014)). Ez a vegyület egy U-U belső hurokhoz kötődik (lenti ábrán zöld rész). Bár ez a vegyület igen specifikus erre a szerkezeti elemre, de egy jó gyógyszertől elvárjuk, hogy csak azt a molekulát gátolja, amely ellen hatni kell, s mást ne (ez a szelektivitás). A feltételezés az, hogy U-U belső hurok sincs túl sok a szervezetben levő funkcionális RNS-ekben, de U-U belső hurok és mellette két bázispárral arrébb egy G-G belső hurok együttes még kevesebb. Tehát a kötés specifikusságát, s így a gyógyszer szelektivitását és hatékonyságát tovább növelendő olyan vegyületet kerestek az adatbázisban, ami a közeli G-G belső hurkot köti. Ez a vegyület egy másik bis-benzimidazol. A két vegyület összekapcsolva elvileg ezt a szerkezeti elemet képes csak kötni. S igen, az így kapott molekula (lenti ábrán 3-assal jelölve) hatékonynak bizonyult az microRNA-96 RNS kötésében és sejttenyészetben a rákos növekedés fékezésében / a rákos sejtek apoptózisának indukálásában.
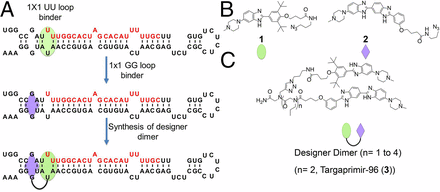
A vegyület RNS-t köt és DNS-t nem. Fontos, hogy más sejtekbe bejutva ott a génállományt ne zavarja. Mutációt indukáló hatása nincs. Egér modellben a vegyület nem mérgező és valamelyest képes visszaszorítani a tumor növekedését (el nem tünteti azt).
Az utolsó pár sorban leírt eredmény a hosszú és drága része a kísérletnek. Minél kevesebb molekulával kell elvégezni, annál olcsóbban jutunk hatékony új gyógyszerhez. Az elejének egy nagy része számítógéppel megoldható, ami sokkal olcsóbb, mint a kísérlet (szaktudás kell hozzá, de amúgy egy sima számítógép elég). Az in silico - számítógépes - technikák egyre fontosabbak a gyógyszerkutatásban. S mostanság az RNS-ek is egyre fontosabb célpontjai a gyógyszerfejlesztésnek, ami számomra is érdekes, mert - bár más okból - de nagyon érdekel az RNS-ek másodlagos szerkezetének és működésének összefüggése.
- Velagapudi, S. P., Cameron, M. D., Haga, C. L., Rosenberg, L. H., Lafitte, M., Duckett, D. R., Phinney, D. G. és Disney, M. D. 2016. Design of a small molecule against an oncogenic noncoding RNA. Proceedings of the National Academy of Sciences 113(21): 5898-5903
- Velagapudi, S. P., Gallo, S. M. és Disney, M. D. 2014. Sequence-based design of bioactive small molecules that target precursor microRNAs. Nature Chemical Biology 10(4): 291-297
- Paul, D. J., Seedhouse, S. J. és Disney, M. D. 2009. Two-dimensional combinatorial screening and the RNA Privileged Space Predictor program efficiently identify aminoglycoside–RNA hairpin loop interactions. Nucleic Acids Research 37(17): 5894-5907
- Velagapudi, S. P., Seedhouse, S. J. és Disney, M. D. 2010. Structure-activity relationships through sequencing (StARTS) defines optimal and suboptimal RNA motif targets for small molecules. Angewandte Chemie (International ed. in English) 49(22): 3816-3818
- Disney, M. D., Labuda, L. P., Paul, D. J., Poplawski, S. G., Pushechnikov, A., Tran, T., Velagapudi, S. P., Wu, M. és Childs-Disney, J. L. 2008. Two-dimensional combinatorial screening identifies specific aminoglycoside−RNA internal loop partners. Journal of the American Chemical Society 130(33): 11185-11194
- Velagapudi, S. P., Seedhouse, S. J., French, J. és Disney, M. D. 2011. Defining the RNA internal loops preferred by benzimidazole derivatives via 2D combinatorial screening and computational analysis. Journal of the American Chemical Society 133(26): 10111-10118
- Aminova, O., Paul, D. J., Childs-Disney, J. L. és Disney, M. D. 2008. Two-dimensional combinatorial screening identifies specific 6′-acylated kanamycin A− and 6′-acylated neamine−RNA hairpin interactions. Biochemistry 47(48): 12670-12679
- Tran, T. és Disney, M. D. 2010. Two-dimensional combinatorial screening of a bacterial rRNA A-site-like motif library: Defining privileged asymmetric internal loops that bind aminoglycosides. Biochemistry 49(9): 1833-1842
- Childs-Disney, J. L., Wu, M., Pushechnikov, A., Aminova, O. és Disney, M. D. 2007. A small molecule microarray platform to select RNA internal loop−ligand interactions. ACS Chemical Biology 2(11): 745-754